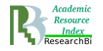Ладутько Е.И.
Институт миркобиологии Национальной академии наук Беларуси,
кандидат биологических наук, ведущий научный сотрудник
Леонович С.И.
Институт миркобиологии Национальной академии наук Беларуси,
младший научный сотрудник
Калинина А.Н
ФГБУ «Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов национального исследовательского центра «Курчатовский институт»,
научный сотрудник
Соколина Л.Н.
РУП «Минская ОСХОС НАН Беларуси»
Заведующая отделом семеноводства зерновых и зернобобовых культур
Заборонок И.М.
РУП «Минская ОСХОС НАН Беларуси»,
директор
Новик Г.И.
Институт миркобиологии Национальной академии наук Беларуси,
кандидат биологических наук,
заведующий лабораторией
Ladutska A.I.
Institute of Microbiology Belarus National Academy of Sciences,
Candidate of Biol. Sci., leading researcher
Leanovich S.I.
Institute of Microbiology Belarus National Academy of Sciences,
Junior researcher
Kalinina A.N.
Federal Institution «State Research Institute of Genetics and Selection of Industrial Microorganisms of the National Research Center» Kurchatov Institute»,
researcher
Sokolina L.N.
Repablican unitary enterprise Regional Agricultural Experiment
Station of the Belarus National Academy of Science,
Head of the Department of seed grain and leguminous crops,
Zaboronok I.M.
Repablican unitary enterprise Regional Agricultural Experiment
Station of the Belarus National Academy of Sciences,
Director
Novik G.I.
Institute of Microbiology Belarus National Academy of Sciences,
Candidate of Biol. Sci., head of laboratory
ISOLATION AND CHARACTERISTICS OF NEW HIGHLY ACTIVE BACTERIAL STRAINS OF THE GENUS PSEUDOMONAS – PROMISING BIOLOGICAL CONTROL AGENTS
ВЫДЕЛЕНИЕ И ХАРАКТЕРИСТИКА НОВЫХ ВЫСОКОАКТИВНЫХ ШТАММОВ БАКТЕРИЙ РОДА PSEUDOMONAS – ПЕРСПЕКТИВНЫХ АГЕНТОВ БИОЛОГИЧЕСКОГО КОНТРОЛЯ
Summary. Ten new strains of bacteria of the genus Pseudomonas, potential biological control agents, were isolated from the potato herb. The method of molecular typing proved strain-specific differences in bacterial cultures. Based on the results of the polymerase chain reaction with the genus-specific primers OpriF and OpriR and phylogenetic analysis, the taxonomic diagnosis of pseudomonads was confirmed. Bacterial cultures are susceptible to bacteriophages and display antifungal activity against phytopathogenic micromycetes of the genera Alternaria and Fusarium. Using the analysis of the nucleotide sequences of the 16S rRNA gene, highly active bacterial strains KaTp1-4, KaTp2-1 and KaTp1-11 were identified as Pseudomonas koreensis and Pseudomonas sp.
Key words: Pseudomonas, molecular-genetic identification, molecular typing, bacteriophages, antifungal activity.
Аннотация. Из травянистой части картофеля выделены десять новых штаммов бактерий рода Pseudomonas – агентов биологического контроля. Методом молекулярного типирования подтверждены штаммоспецифические различия бактериальных культур. На основании результатов полимеразной цепной реакции с родоспецифичными праймерами OpriF и OpriR и филогенетического анализа подтвержден таксономический диагноз псевдомонад. Культуры бактерий обладают чувствительностью к бактериофагам и антифунгальной активностью в отношении фитопатогенных микромицетов родов Alternaria и Fusarium. С помощью анализа нуклеотидных последовательностей гена 16S рРНК высокоактивные штаммы бактерий KaTp1-4, KaTp2-1 и KaTp1-11 идентифицирован как Pseudomonas koreensis и Pseudomonas sp.
Ключевые слова: Pseudomonas, молекулярно-генетическая идентификация, молекулярное типирование, бактериофаги, антифунгальная активность.
Введение. Картофель является одной из основных сельскохозяйственных культур. Известно, что в период вегетативного размножения картофеля наблюдается присутствие возбудителей болезней на ботве в период вегетации культур и в клубнях во время хранения. Рост устойчивости возбудителей к химическим средствам защиты растений и постоянные вспышки заболеваний приводят к увеличению применяемых доз химических препаратов, что, в свою очередь, индуцирует новое повышение устойчивости патогенов. Поэтому для повышения эффективности растениеводства актуальны подходы, предполагающие использование биологических средств защиты на основе микроорганизмов — естественных обитателей экосистемы [1], которые обладают избирательностью действия и позволяют избежать нежелательных изменений в биоценозах, а также сохранить полезные свойства продукции. Большинство таких микроорганизмов синтезируют различные метаболиты, индуцирующие в растении каскад защитных реакций [2]. Кроме того, при взаимодействии с растением бактерии могут повышать его жизнеспособность, тем самым регулируя численность фитопатогенов. В результате формируются несколько уровней защиты, включающих прямую конкуренцию с фитопатогеном, продукцию биопленок и антибиотических веществ, а также индукцию собственного иммунитета растения. Особый интерес представляют эндофитные бактерии [3], обитающие в растительных тканях, что делает их менее зависимыми от внешних факторов. Эндофиты с комплексом хозяйственно полезных признаков могут способствовать формированию длительной защиты растения от стрессовых факторов [4]. Бактерии рода Pseudomonas – одна из наиболее изученных групп микроорганизмов с точки зрения объектов биологического контроля бактериальных и грибных заболеваний сельскохозяйственных культур. Механизмы антагонистического взаимодействия псевдомонад и фитопатогенов включают способность к синтезу широкого спектра антибиотических и ростстимулирующих веществ, конкуренцию за источник питания, эффективную колонизацию филлосферы [5–7]. В связи с большой востребованностью биопрепаратов, актуален вопрос точной идентификации входящих в их состав микроорганизмов. Для устранения недостатков классических фенотипических методов идентификации за последние несколько лет созданы молекулярные методы таксономической идентификации бактерий различных таксономических групп, в том числе рода Pseudomonas. Развитие методов на основе полимеразной цепной реакции (ПЦР) открыло новые возможности для четкой и быстрой идентификации псевдомонад [8]. Удобные в исполнении методики на основе ПЦР являются альтернативой классическим микробиологическим методам и предпочтительны в случае необходимости немедленного получения результата. Молекулярно-генетические методы позволяют решать задачи индикации, относящиеся к указанному роду, и точной видовой идентификации.
Целью данной работы явилось выделение из травянистой части картофеля новых высокоактивных штаммов бактерий рода Pseudomonas перспективных агентов биологического контроля, таксономическая характеристика и молекулярно-генетическая идентификация культур.
Материалы и методы. Для выделения бактерий 5 г исследуемого растительного материала, предварительно промытого дистиллированной водой, измельчали и смешивали со стерильными порциями мясо-пептоного бульона (МПБ), после чего делали ряд десятикратных разведений. Бактериальную суспензию (100 мкл) из разведений 10-3 – 10-7 высевали на МПБ, культивировали при 28°С в течение 24 – 48 ч. Изолированные колонии рассевали на мясо-пептонном агаре (МПА) до получения монокультур (не менее трех пассажей). Чистоту культуры контролировали визуально и микроскопически. Изолированные бактерии культивировали на МПА и МПБ при 28°С в течение 24 – 48 ч; мицелиальные грибы – на картофельно-глюкозной жидкой среде при комнатной температуре в течение 72 ч. Проверка антифунгальной активности производилась методом лунок [9]. В качестве тест культур использованы мицелиальные грибы из коллекционного фонда Белорусской коллекции непатогенных микроорганизмов (БКМ): Fusarium graminearum БИМ F-601, Fusarium proliferatum БИМ F-602, Fusarium sp. БИМ F-603, Alternaria brassicae БИМ F-621. Морфологию клеток исследовали методом световой микроскопии, используя микроскоп Nicon Eclipse E2000 (Nicon corporation, Япония). При микроскопии клеток использовали окрашивание по методу Грама [10]. Идентификацию микроорганизмов проводили на основе изучения морфологических, культуральных, физиолого-биохимических свойств выделенных микроорганизмов в соответствии с Определителем бактерий Берджи (1997), а также при помощи молекулярно-генетического метода идентификации на основании анализа нуклеотидной последовательности гена 16S рРНК.
Хромосомную ДНК бактерий выделяли при помощи наборов реагентов «Jena» (Германия). Для проведения ПЦР использовали праймеры и реагенты производства «Праймтех» (Беларусь). Амплификацию нуклеотидной последовательности гена 16S рРНК проводили с универсальными эубактериальными праймерами 8f (5ʹ-agagtttgatcctggctcag-3ʹ) и 1492r (5ʹ-ggttaccttgttacgactt-3ʹ), используя температурно-временной профиль: 95°С – 3 мин (1 цикл); 95°С – 30 с, 57°С – 30 с, 72°С – 1,5 мин (35 циклов); 72°С – 5 мин (1 цикл). В качестве стандартов для определения размера продуктов ПЦР применяли маркер молекулярной массы фрагментов Gene Ruler DNA Ladder 1Kb Plus («Fermentas», Литва).
Для очистки амплифицированных фрагментов гена 16S рРНК использовали набор DNA Extraction kit («Thermo Scientific», Литва). Реакцию секвенирования ПЦР-фрагментов гена 16S рРНК проводили на автоматическом секвенаторе АЕ3000 с использованием набора реагентов для секвенирования BigDye Terminator Cycle Sequencing Kit («Applied Biosystems», США). Для анализа результатов секвенирования и редактирования последовательностей применяли программу eSeq. Сравнительный анализ секвенированных фрагментов генов выполняли с использованием программы BLAST базы данных GenBank (http://www.ncbi.nlm.nih.gov/blast.).
Для проведения молекулярного типирования геномной ДНК использовали праймеры М13 (5’-gagggtggcggttct-3’) и 1254 (5’-ccgcagccaa-3’). Амплификацию проводили в оптимизированных условиях на автоматическом термоциклере Mastercycler®pro (Eppendorf) и SureCycler 8800 Thermal Cycler (Agilent Technologies). В работе использовали реагенты и праймеры производства «Праймтех» (Беларусь). На один образец готовили реакционную смесь, содержащую буфер АМ (х10) для Taq-полимеразы (2 мкл), 0,25 мкл. Taq-полимеразы, 1 мкл каждого праймера (при концентрации 10 pmol/µl), 1 мкл ДНК-матрицы и бидистиллированную воду (15,75 мкл). Общий объем реакционной смеси составлял 20 мкл. Реакцию амплификации c праймером М13 проводили, используя следующий температурно-временной профиль: денатурация 3 мин при 95ºС, 39 циклов элонгации – 95ºС – 30 с, 45ºС – 30 с, 72ºС – 2 мин, достройка цепи 5 мин при 72ºС; охлаждение до 4ºС. Для реакции амплификации c праймером 1254: денатурация 3 мин при 95ºС, 39 циклов элонгации – 95ºС – 30 с, 45ºС – 30 с, 72ºС – 1 мин, достройка цепи 5 мин при 72ºС; охлаждение до 4ºС.
Амплификацию нуклеотидной последовательности гена 16S рРНК проводили с использованием родоспецифичных праймеров OpriF (5’-atgaacaacgttctgaaattctctgct-3’) и OpriR (5’-cttgcggctggctttttccag-3’). Реакционная смесь обьемом 20 мкл содержала 1 мкл ДНК-матрицы (10-20 нг), 2 мкл 10Х “АМ” буфера для Taq-полимеразы, 2 мкл смеси дНТФ (по 2 мМ), по 1 пМ каждого праймера, 0,25 мкл Taq-полимеразы (1,25 ед.). Температурно-временной профиль амплификации: 1 цикл – 5 мин при 95 ºС; 30 циклов – 95 ºС – 30 сек, 60 ºС – 30 сек, 72 ºС – 20 сек; 1 цикл – 5 мин при 72 ºС; Охлаждение до 4ºС.
Образцы ДНК и продукты ПЦР анализировали методом электрофореза в 1,5 % агарозном геле с использованием 1×ТАЕ-буфера при напряженности электрического поля 5 В/см в течении 45 мин. Для визуализации ДНК гель окрашивали раствором бромистого этидия в концентрации 0,05 мкг/мл. В качестве стандарта для определения размера продуктов ПЦР применяли маркер молекулярной массы ДНК O’GeneRuler Mix DNA Ladder (ThermoScientific). Образцы по 3 мкл смешивали с 1 мкл красителя 6х Orange 2D (ThermoScientific) и далее 3 мкл смеси загружали в лунки агарозного геля.
При предварительном анализе нуклеотидной последовательности гена 16S рРНК использовали программы BLAST. Далее нуклеотидную последовательность 16S рДНК изучаемого изолята сравнивали с нуклеотидными последовательнсотями типовых штаммов близкородственных видов с помощью программы CLUSTAL W [11] и корректировали. Построение филогенетического древа производили с помощью пакета программ MEGA6 (Molecular Evolutionary Genetics Analysis) (http://www.megasoftware.net/) с использованием метода Neighbor-Joining Bootstrap. Статистическую достоверность ветвления оценивали с использованием соответствующей функции программы MEGA6 на основе 1000 альтернативных деревьев.
Результаты. Из листьев и стеблей картофеля выделены штаммы грамотрицательных бактерий, обладающих чувствительностью к бактериофагам и антифунгальной активностью в отношении фитопатогенных грибов родов Alternaria, Fusarium [12]. Десять штаммов бактерий были идентифицированы как представители рода Pseudomonas. На поверхности агаризованных питательных сред псевдомонады образовывали круглые, гладкие, непрозрачные колонии бледно-желтого цвета. При микроскопическом исследовании нативных и фиксированных препаратов клеток характерны грамотрицательные палочки, подвижные за счет наличия полярных жгутиков. Палочки аэробные, каталазо- и оксидазоположительные, что характерно для представителей рода Pseudomonas.
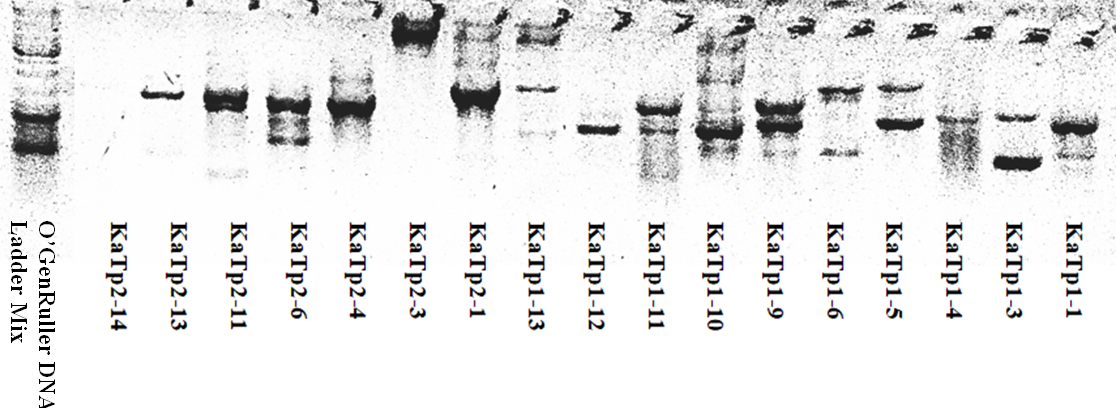
Для анализа генетической геторогенности исследуемых штаммов проведена сравнительная характеристика культур с использованием метода RAPD-ПЦР (рис.1).
А
Б
Рисунок 1 .Электрофореграмма продуктов RAPD-ПЦР бактериальных изолятов.
А — праймер М13, Б — праймер 1254
При использовании праймера 1254 для анализа генетической гетерогенности псевдомонад получены штаммоспецифичные RAPD-профили, содержащие от трех до двенадцати ампликонов размером ~ 200-3500 п.н. При проведении RAPD-ПЦР с праймером M13 число ПЦР-фрагментов в фингерпринтах варьирует от шести до двенадцати, размером ~ 250-9000 п.н. Электрофоретические профили характеризовались специфическими индивидуальными ампликонами, отличались по количеству и размеру ПЦР-продуктов, что позволило их дифференцировать и выявить внутривидовую генетическую гетерогенность бактериальных штаммов.
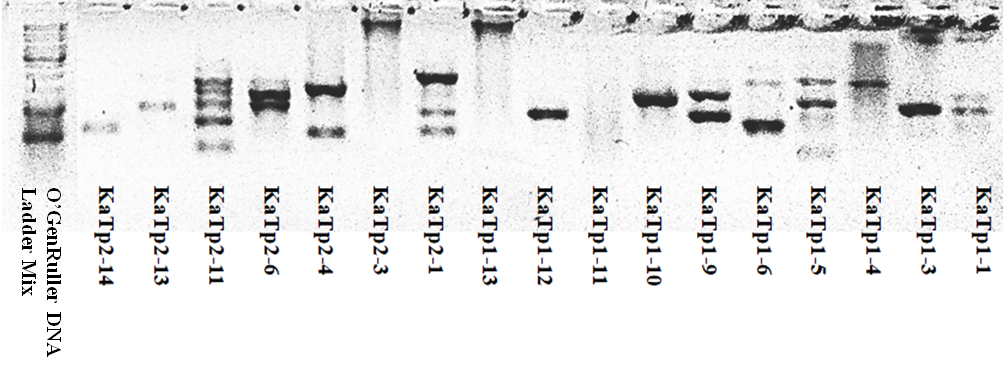
Для подтверждения принадлежности культур бактерий к роду Pseudomonas были выбраны родоспецифичные праймеры OpriF и OpriR, амплифицирующие участок гена липопротеина I у бактерий рода Pseudomonas и дающих ампликон размером 249 п.н. [8]. Используя сервер NCBI Primer Blast и базу данных GenBank, была подтверждена специфичность данных праймеров к роду Pseudomonas и экспериментально доказана на идентифицированных (на основе анализа гомологии нуклеотидной последовательности гена 16S рРНК и референтных последовательностей баз данных GenBank и Ribosomal Database Project (RDP)) культурах из Белорусской коллекции непатогенных микроорганизмов. В качестве отрицательного контроля использовали коллекционные культуры бактерий видов Arthrobacter arilatensis, Chryseobacterium nakagawai, Bacillus licheniformis, Bacillus cereus, Bacillus subtilis, в качестве положительного контроля коллекционные культуры бактерий Pseudomonas koreensis, Pseudomonas argensinensis, Pseudomonas extremorientalis и Pseudomonas asplenii. Полученные ампликоны представлены на рисунке 2.
Рисунок 2. – Электрофореграмма продуктов амплификации фрагмента ДНК гена 16S рРНК коллекционных культур бактерий с использованием праймеров OpriF и OpriR:
б/н слева – маркер молекулярной массы ДНК O’GeneRuler DNA Ladder Mix; 1- отрицательный контроль; 2- Pseudomonas asplenii; 3- Pseudomonas extremorientalis; 4 — Bacillus subtilis; 5- Bacillus cereus; 6 — Bacillus licheniformis;7 — Pseudomonas argensinensis; 8 — Chryseobacterium nakagawai; 9 — Pseudomonas koreensis; 10 – Arthrobacter arilatensis;
В результате амплификации праймерами OpriF и OpriR были получены ампликоны размером около 269 п.н. коллекционных бактерий рода Pseudomonas, что соответствует размеру участка гена липопротеина I.
Проведена ПЦР образцов ДНК изолятов бактерий, выделенных из листьев и стеблей картофеля, с использованием родоспецифичных праймеров OpriF и OpriR. Электрофореграмма представлена на рисунке 3. В качестве отрицательного контроля были использованы образцы ДНК бактериальных изолятов, выделенных из листьев и стеблей картофеля. Бактерии идентифицированы нами ранее как представители родов: Arthrobacter, Bacillus, Chryzeobacterium, Pantoea, Stenotrophomonas на основании анализа данных нуклеотидных последовательностей гена 16S рРНК. По результатам ПЦР-анализа с родоспецифичными праймерами OpriF и OpriR культуры: KaTp2-14, KaTp2-13, KaTp2-11, KaTp2-6, KaTp2-1, KaTp1-12, KaTp1-11, KaTp1-10, KaTp1-9, KaTp1-4 являются представителями рода Pseudomonas.
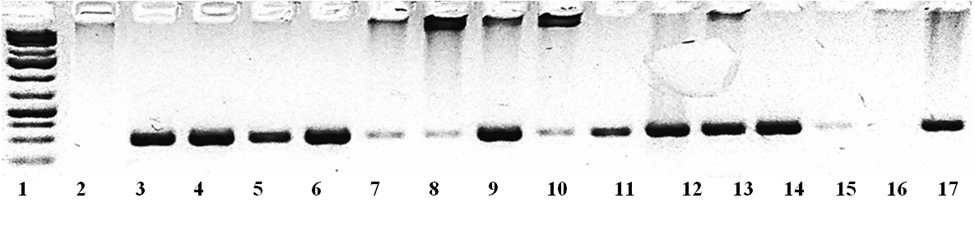
Рисунок 3 – Электрофореграмма продуктов амплификации фрагмента ДНК гена 16S рРНК бактериальных культур, выделенных из листьев и стеблей картофеля с использованием праймеров OpriF и OpriR.
№1 — маркер молекулярной массы ДНК O’GeneRuler DNA Ladder Mix (ThermoScientific), № 2 — Bacillus thuringiensis, № 7 — Arthrobacter arilaitensis, № 8 — Stenotrophomonas rhizophila, № 10 — Stenotrophomonas rhizophila, № 15 — Chryseobacterium oncorhynchi, № 16 — Pantoea agglomerans; изоляты: № 3-KaTp2-14, №4-KaTp2-13, №5- KaTp2-11, №6 — KaTp2-6, №9 — KaTp2-1, №11 — KaTp1-12, № 12 — KaTp1-11, № — 13 KaTp1-10, № 14 — KaTp1-9, № 17 — KaTp1-4.
Для высокоактивных штаммов KaTp2-1, KaTp1-11 и KaTp1-4, обладающих антагонистической активностью, определены нуклеотидные последовательности гена 16S рРНК. Филогенетический анализ нуклеотидных последовательностей гена 16S рРНК показал, что штаммы являются типичными представителями рода Pseudomonas. На филогенетическом древе исследуемые штаммы распределены в разные подгруппы псевдомонад: штамм KaTp2-1 находится в одной подгруппе с представителями бактерий вида Pseudomonas argentinensis. Штамм KaTp1-11 занимает положение в подгруппе с представителями бактерий вида Pseudomonas koreensis (рис. 4). Точная выдовая идентификация данных штаммов затруднительна из-за малой длины секвенированного фрагмента.

Рисунок 4. Филогенетическое древо, построенное с использованием метода «Neighbor-Joining», основанное на сравнении нуклеотидных последовательностей 16S рДНК видов рода Pseudomonas, близкородственных исследуемым штаммам. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью «bootstrap»-анализа 500 альтернативных деревьев.
Штамм KaTp1-4 отнесен в отдельную подгруппу представителей бактерий вида Pseudomonas koreensis. Исследуемые участки ДНК штамма KaTp1-4 на 99,9% схожи с геном 16S рРНК штамма Pseudomonas koreensis KJ937669.1 из базы данных GenBank. По результатам молекулярно-генетического исследования штамм KaTp1-4 идентифицирован как Pseudomonas koreensis, а два других штамма: KaTp2-1 и KaTp1-11 как Pseudomonas sp. соответственно.
Исследована антифунгальная активность выделенных штаммов псевдомонад в отношении фитопатогенных грибов родов Alternaria и Fusarium. Результаты исследований представлены в таблице 1. Установлено, что штамм KaTp1-4 является антагонистом фитопатогенных грибов родов Alternaria и Fusarium. Штаммы KaTp1-11 и KaTp2-1 проявляют антифунгальную активность в отношении грибов рода Fusarium.
Таблица 1 – Ингибирование роста и развития микромицетов бактериями-антагонистами
| Изолят | Штамм микромицетов | |||
| Fusarium graminearum
БИМ F-601 |
Fusarium proliferatum
БИМ F-602 |
Fusarium sp.
БИМ F-603 |
Alternaria brassicae
БИМ F-621 |
|
| KaTp1-4 | IS / * | IS / ** | IR / *** | IR / ** |
| KaTp1-11 | IR / ** | – | – | – |
| KaTp2-1 | – | IS / ** | – | – |
Примечание: * – зона ингибирования роста фитопатогенных мицелиальных грибов лежит в диапазоне 10-30 мм; ** – 40-60 мм; *** – 70-100 мм; IR – изолят ингибирует рост мицелия; IS – изолят ингибирует спорообразование фитопатогенов.
Выявлено, что штам KaTp1-4 ингибирует процесс спорообразования и рост мицелия микромицетов родов Fusarium и Alternaria. Штаммы KaTp1-11 и KaTp2-1 подавляют рост и развитие грибного мицелия.
Таким образом, из травянистой части картофеля выделены десять штаммов бактерий рода Pseudomonas. При помощи молекулярного типирования подтверждены штаммоспецифические различия бактериальных культур. На основании ПЦР с родоспецифичными праймерами и филогенетического анализа подтвержден их таксономический диагноз. Три культуры бактерий обладают антифунгальной активностью в отношении фитопатогенных мицелиальных грибов. Штамм KaTp1-4 идентифицирован как Pseudomonas koreensis, а два других: KaTp2-1 и KaTp1-11 как Pseudomonas sp.
Литература.
- Bale J.S., van Lenteren J.S., Bigler F. Biological control and sustainable food production. Phil. Trans. R. Soc. B. 2008. Vol. 363. № 1492. P. 761-776.
- Rosenblueth M., Martínez-Romero E. Bacterial endophytes and their interactions with hosts. Mol. Plant-Microbe Interact. 2006. Vol. 19. P. 827-837.
- Schulz B., Boyle C. T.N. Sieber (eds.). What are endophytes? In: Microbial root endophytes. Springer-Verlag, Berlin. 2006. P. 1-13.
- Maksimov I.V., Abizgl’dina R.R., Pusenkova L.I. Plant growth promoting rhizobacteria as alternative to chemical crop protectors from pathogens (review). Prikladnaya biokhimiya i mikrobiologiya. 2011. Vol. 47. P. 373-385
- Боронин А.М., Кочетков В.В. Биологические препараты на основе псевдомонад. АГРО XXI. 2000. 140 с.
- Штерншис М.В., Джалилов Ф.С., Андреева И.В., Томилова О.Г. Биопрепараты в защите растений. Новосибирск. 2000. 128 с.
- Benizri E., Baudon E., Guckert A. Root colonization by inoculated plant growth-promoting rhizobacteri. Biocontrol science and technology. 2001. № 11. P. 557–574.
- Gholami A., Majidpour A., Talebi-Taher M. PCR-based assay for the rapid and precise distinction of Pseudomonas aeruginosa from other Pseudomonas species recovered from burns patients. J PREV MED HYG. 2016. Vol.57. P. 81-85.
- Методы почвенной микробиологии / Сэги Й. – М.: Колос, 1983. ‑ 253с.
- Нетрусов, А.И. .А. Егорова, Л.М. Захарчук. Практикум по микробиологии / Москва: Академия. 2005. – 608 с.
- J.D. Thompson, D.G. Higgins, T.J. Gibson CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice. Nucleic Acids Res. 1994. Vol. 22. P. 4673–4680.
- Леонович С.И., Калинина А.Н., Кантерова А.В., Ладутько Е.И., Новик Г.И. Изоляция и характеристика бактерий, обладающих антифунгальной активностью в отношении фитопатогенных грибов родов Rhizoctonia, Fusarium, Alternaria. Сб. науч. трудов Инститата микробиологии НАН Беларуси «Микробные биотехнологии: фундаментальные и прикладные аспекты» Т. 10. 2018. С. 98-106.